陈巍:蛋白质求解网络AlphaFold2 —《AI芯片设计:原理与实践》节选
《AI芯片设计:原理与实践》系列,适合AI芯片设计人员入门与芯片赛道投资人了解技术内涵。系列目录附在本文最后,本文介绍第3.10节蛋白质求解网络AlphaFold2 。
■ 陈巍,资深芯片专家,人工智能算法-芯片协同设计专家,擅长芯片架构与存算一体。
■ 耿云川,资深SoC设计专家,软硬件协同设计专家,擅长人工智能加速芯片设计。
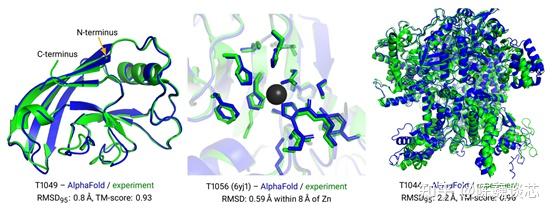
DeepMind(Google旗下)的AlphaFold2在蛋白质结构预测大赛CASP 14中,对大部分蛋白质结构的预测与真实结构只差一个原子的宽度,达到了人类利用冷冻电镜等复杂仪器检测的水平。这是蛋白质结构预测技术上的巨大进步,并被Nature和Science选为2021年度十大科学突破。
折叠的蛋白质可以被视为氨基酸的“空间图”,其中残基是结点或端点,边线将残基紧密相连。蛋白质结构预测是生物信息学与理论化学所追求的重要目标之一,解析蛋白质的空间结构对于认识蛋白质的功能、功能的执行、生物大分子间的相互作用,以及医学和药学的发展(如药物靶点的设计等)具有重要意义。
根据不同的氨基酸和序列,蛋白质能折叠成的构型数量是一个天文数字,因此很难用常规方法进行蛋白质结构的准确预测。例如,实验的方法(例如冷冻电镜)至今才能解出10万的蛋白质结构,但是氨基酸序列有10亿的序列,传统的实验方法解出一个蛋白的结构可能需要花费数月甚至数年的时间。
在CASP14上,DeepMind直接总分92.4,和实验的误差在1.6 埃,即使是在最难的没有同源模板的蛋白质上面,这个分数也达到了了令人震惊的87.0。AlphaFold2得分远远超过第二的成绩。第一名和第二名之间的差距,甚至比第二名到最后一名的差距还要大。
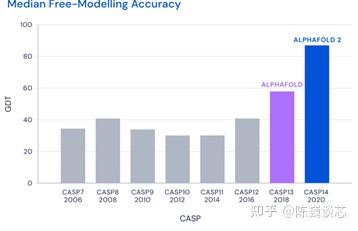
蛋白质结构预测大赛CASP(Critical Assessment of protein Structure Prediction)用来衡量预测准确性的主要指标是 GDT,范围为0-100。简单来说,GDT可以近似地认为是和实验结构相比,成功预测在正确位置上的分数。70分就是达到了同源建模的精度,大约90 分可以和实验结果相竞争。
AlphaFold2这类模型是深度学习算法历史上的里程碑,将为人类的健康和医疗做出巨大贡献。
3.10.1 蛋白质结构预测
蛋白质结构:是指蛋白质分子的空间结构。蛋白质是生命活动的体现者,蛋白质的结构决定了蛋白质的生物学功能。由线性氨基酸组成的蛋白质需要折叠(Fold)成特定的空间结构才具有相应的生理活性和生物学功能。
蛋白质结构预测(Protein Structure Prediction):是指从蛋白质的氨基酸序列中预测蛋白质的三维结构。也就是说,从蛋白质的一级结构预测其折叠和二级、三级、四级结构。
残基(Residue):蛋白质都是由20种不同的L型α氨基酸连接形成的多聚体,在形成蛋白质后,这些氨基酸就是残基。之所以被称为残基,是由于在蛋白质的序列中,组成多肽的氨基酸在相互结合时,其部分基团形成肽键而失去水分子(氨基酸之间的氨基和羧基脱水成键),剩余的结构部分就是氨基酸残基。
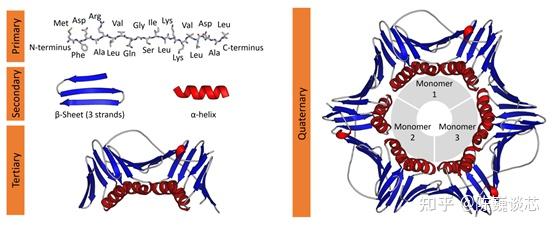
蛋白质的分子结构可划分为四级,以描述其不同层级的特征:
蛋白质一级结构:组成蛋白质多肽链的线性氨基酸序列。
蛋白质二级结构:依靠不同氨基酸之间的C=O和N-H基团间的氢键形成的稳定结构,主要为α螺旋和β折叠。
蛋白质三级结构:通过多个二级结构元素在三维空间的排列所形成的一个蛋白质分子的三维结构。
蛋白质四级结构:用于描述由不同多肽链(亚基)间相互作用形成具有功能的蛋白质复合物分子。
蛋白质结构预测理论基础
蛋白质的高级结构由其一级结构序列决定,蛋白质可以自发地折叠成它们的天然结构,特别是结构简单的蛋白质小分子。
序列相似的蛋白质具有相似的三维结构,且测定蛋白质序列比测定蛋白质结构容易得多,而蛋白质结构可以给出比序列更多的关于其功能机制的信息。
3.10.2 AlphaFold2特点
AlphaFold 通过结合基于蛋白质结构的进化、物理和几何约束的新型神经网络架构和训练程序,极大地提高了结构预测的准确性。
AlphaFold2的主要特点包括:
1) AlphaFold2广泛使用了注意力(Attention)机制。经过端到端训练,使用注意力机制分析和解析氨基酸连接图的结构,同时对所构建的隐式图进行三维结构推理。特别是在注意力机制中分别按行和按列进行正交化的分析,降低了计算量。
2) 使用欧几里得空间来表征蛋白质结构,进而通过循环式的自注意力机制获得预测结构的解空间收敛。
3.10.3 AlphaFold2网络结构
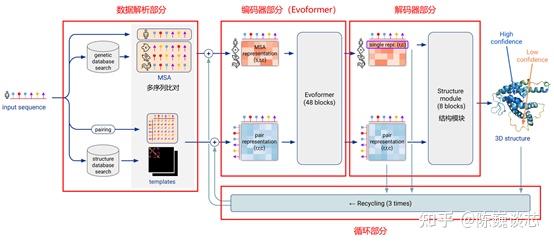
AlphaFold的详细结构比较复杂,在本文主要介绍该模型的基本结构与思路。
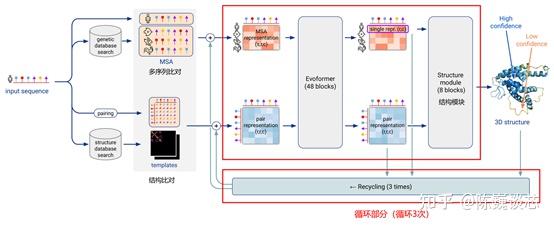
AlphaFold2模型架构可以分为四部分:数据解析部分、编码器部分(Evoformer)、解码器部分(Structure Module)、循环部分(Recycling)。
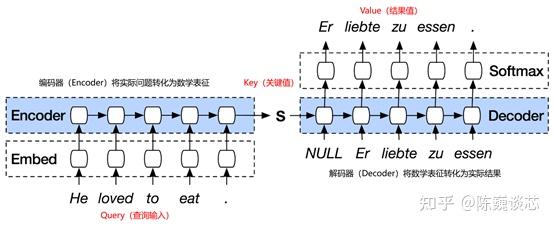
模型输入为待预测的蛋白质序列,输出为预测的蛋白质三维结构。这与BERT等NLP模型通过计算Query(查询输入)与Key(关键值)的关联度,生成最合适的Value(结果值)的思路非常接近。
3.10.3.1 数据解析部分
数据解析部分用于输入序列和数据库的解析,为编码器提供输入。
在这一部分,使用了氨基酸序列数据库和结构数据库,分别用于相近序列的比对和结构模板的配对。
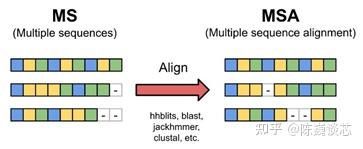
生物信息学的基础是基于这样的一个假设:序列相似,结构相似,功能相似。一般认为相近的序列或者相近的结构会衍生出相近的功能域。
1)序列数据库被用于多序列比对(Multiple Sequence Alignment,MSA),即在序列数据库中检索与输入序列接近的数据库序列。
2)结构数据库则用于结构匹配,寻找与输入序列的结构接近的已知结构模板。
然后序列比对与结构比对的结果作为输入传输给编码器部分。
3.10.3.2 编码器部分 (Evoformer)
解码器部分即Evoformer,一共48层,每层不共享权重。
序列比对以MSA表征的方式传入编码器部分,三维矩阵的s、r、c分别表示序列的数量,比对氨基酸的数量和通道数。
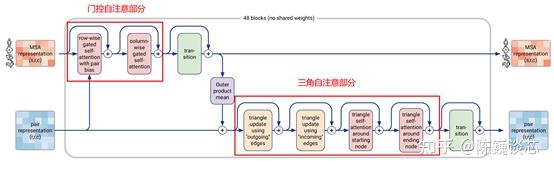
MSA表征数据进入48层的Evoformer,每个Evoformer层可以划分为门控自注意部分和三角自注意部分。
1) 在门控自注意部分中,进行序列与氨基酸关系的匹配。MSA依次通过按行门控自注意和按列门控自注意。即对矩阵先按行再按列做Attention,通过局部结构正交做Attention,不断优化Embedding的结构。按行Attention就是每个氨基酸序列里的Self-Attention,按列Attention则是与其他氨基酸的匹配关系。这里的门控与LSTM的输出门原理一样,通过训练后的门控来控制不同元素的输出或者不输出,进而控制序列和氨基酸的对应关系。
2) 在三角自注意部分中,进行氨基酸结构关系的匹配。这里是通过4种端点三角形更新模式进行的。这些更新模式可以根据初始的氨基酸匹配数据和序列-氨基酸匹配数据推导(或训练)出三维空间的氨基酸关联,通过边线和端点的连接匹配完成三维空间的氨基酸结构匹配。
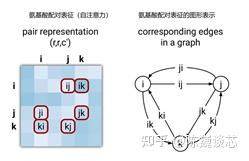
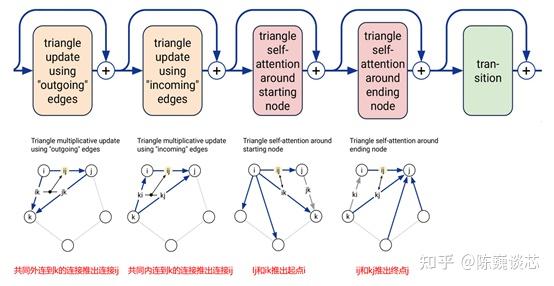
3.10.3.3 解码器部分(Structure Module )
由于用绝对位置编码蛋白质的三维坐标的表征不唯一,且对于旋转或者位移等坐标变化很不友好,因此有必要用相对位置来编码蛋白质结构的三维坐标。解码器部分一共8层,共享权重,这部分类似于传统的RNN模型。
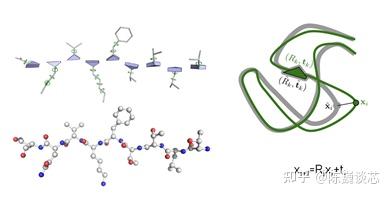
在这里用下一个氨基酸相对于上一个氨基酸的位置变换向量来表征蛋白质结构。
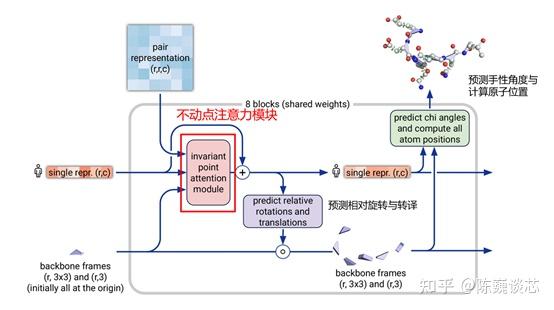
结构模块使用MSA中的第一条(即只保留目标氨基酸序列,丢弃剩余部分,称为single repr)和前面Evoformer计算得到的匹配特征。
不动点注意力模块是基于欧几里得变换的注意力机制。在欧几里得变化下,全局序列结构是不变的。通过不动点注意力模块可实现相应的坐标转换,进而完成相对旋转和手性角度的预测。
3.10.3.4 循环部分(Recycling)
在AlphaFold2模型中,编码器和解码器整体循环3次(类似于函数迭代),逐级数据修正,以获得更高精度的序列预测结果。
参考文献
Jumper, J. , et al. "Highly accurate protein structure prediction with AlphaFold." Nature (2021):1-11.
附:《AI芯片设计:原理与实践》 目录
1 概述
1.1 AI芯片的应用场景
1.2 AI芯片的分类
1.3 AI芯片设计基本流程
Reference
2 AI芯片常用机器学习模型
2.1 线性模型
2.2 SVM
2.2.1 线性分类器及其求解
2.2.2 核函数
2.2.3 松弛变量
2.3 贝叶斯网络
2.3.1 贝叶斯定理
2.3.2 贝叶斯网络结构
Reference
3 AI芯片常用深度学习模型
3.1 基础知识
3.1.1 全连接
3.1.2 卷积
3.1.3 激活函数
3.2 图像分类网络AlexNet 25
3.2.1 AlexNet特点
3.2.2 AlexNet网络结构
3.3 ResNet
3.3.1 图像分类网络ResNet特点
3.3.2 ResNet网络结构
3.3.3 ResNet算法细节
3.4 目标检测网络Faster-RCNN
3.4.1 RCNN与Fast-RCNN
3.4.2 Faster-RCNN特点
3.4.3 Faster-RCNN算法结构
3.5 目标检测网络SSD
3.5.1 SSD特点
3.5.2 SSD网络结构
3.5.3 SSD算法细节
3.6 目标检测网络YOLOv5
3.6.1 YOLOv5特点
3.6.2 YOLOv5网络结构
3.6.3 YOLOv5算法细节
3.7 自然语言处理网络BERT
3.7.1 BERT特点
3.7.2 Attention机制
3.7.3 Transformer
3.7.4 Bert网络结构
3.7.5 Bert算法细节
3.8 图像分类网络SqueezeNet
3.8.1 SqueezeNet特点
3.8.2 SqueezeNet网络结构
3.9 图像分类网络MobileNet
3.9.1 MobileNet特点
3.9.2 MobileNet网络结构
3.10 蛋白质求解网络AlphaFold2
3.10.1 AlphaFold2特点
3.10.2 AlphaFold2网络结构
3.10.3 AlphaFold2算法细节
Reference
4 AI加速电路设计
4.1 卷积计算加速
4.1.1 传统MAC阵列设计
4.1.2 基于存算一体技术的MAC阵列设计
4.1.3 数据-电路映射结构
4.2 池化加速
4.3 激活函数加速
4.4 Softmax加速
4.5 BatchNormal加速
5 GPU架构
5.1 基本架构
5.2 NVDLA解读
5.2.1 两种实现模型的比较
5.2.2 NVDLA结构
5.2.3 NVDLA主要模块
Reference
6 DSA架构
6.1 基本架构
6.2 谷歌TPU
6.3 海思达文西
6.4 Wave Computing
Reference
7 可重构架构
7.1 传统可重构
7.2 软件定义的可重构
7.3 DSP电路结构
Reference
8 存算一体架构
8.1 存算一体架构的分类
8.2 近存计算
8.3 基于SRAM/DRAM的存内计算
8.4 基于FLASH的存内计算
8.5 基于新型NVM的存内计算
8.6 内存逻辑
8.7 可重构存算
Reference
9 适用AI芯片的量化与稀疏化技术
9.1 量化技术
9.2 稀疏化技术
Reference
10 编译器设计
10.1 主流AI编译器概况
10.2 专用编译器设计流程
10.3 通用AI编译器设计流程
10.4 泛AI编译器设计流程
Reference
11 AI芯片的未来
11.1 AI芯片应用与落地趋势
11.2 AI芯片架构发展趋势
11.3 AI芯片市场发展现状与趋势
Reference
12 基于FPGA的图像分类AI加速器设计实战
12.1 架构设计实战
12.2 MAC阵列设计实战
12.3 Softmax加速设计实战
12.4 数据流-存储-计算协同优化实战





